| 生命短暂,艺术长流——水生所深入探究蓝细菌RNA降解调控机制 |
| 文章来源: | 发布时间:2022-04-12 | 【打印】 【关闭】 | 浏览:次 |
|
"Life is short, art is long (生命短暂,艺术长流)." RNA半衰期很短,但其行使的功能却无比重要,人们对其探索也永无止境。这点正体现了古希腊伟大医学家希波克拉底(Hippocrates)上述名言的精髓。
RNA不但介导了遗传信息的解码过程,还在一系列生命过程中起调控和结构功能。中国科学院水生生物研究所张承才研究员团队与德国Wolfgang R. Hess教授团队合作的文章"'Life is short, and art is long': RNA degradation in cyanobacteria and model bacteria"于3月24日在国内高起点新刊物、微生物学综合性期刊mLife创刊号上在线发表。该文讨论了蓝细菌和模式菌中RNA代谢机制,揭示了不同细菌RNA代谢的共通性和多样性。作该本领域的首篇综述,文章可为后续相关研究提供重要参考。 RNA在细胞内种类繁多,它们既是遗传信息的载体(mRNA),也是蛋白翻译机器的组分(rRNA、tRNA),还直接发挥基因调控的功能(asRNA、sRNA等)。与其他大分子相比,RNA代谢迅速,平均半衰期仅几分钟。不同细菌中的mRNA降解过程基本相同,涉及三类酶:RNA内切酶,RNA外切酶,以及一些起辅助功能的酶(包括RNA焦磷酸水解酶、RNA解旋酶等)(图1)。催化不同RNA降解步骤的RNA酶还能通过形成酶复合体(即RNA降解体)等形式协作,以确保RNA降解能够高效进行。
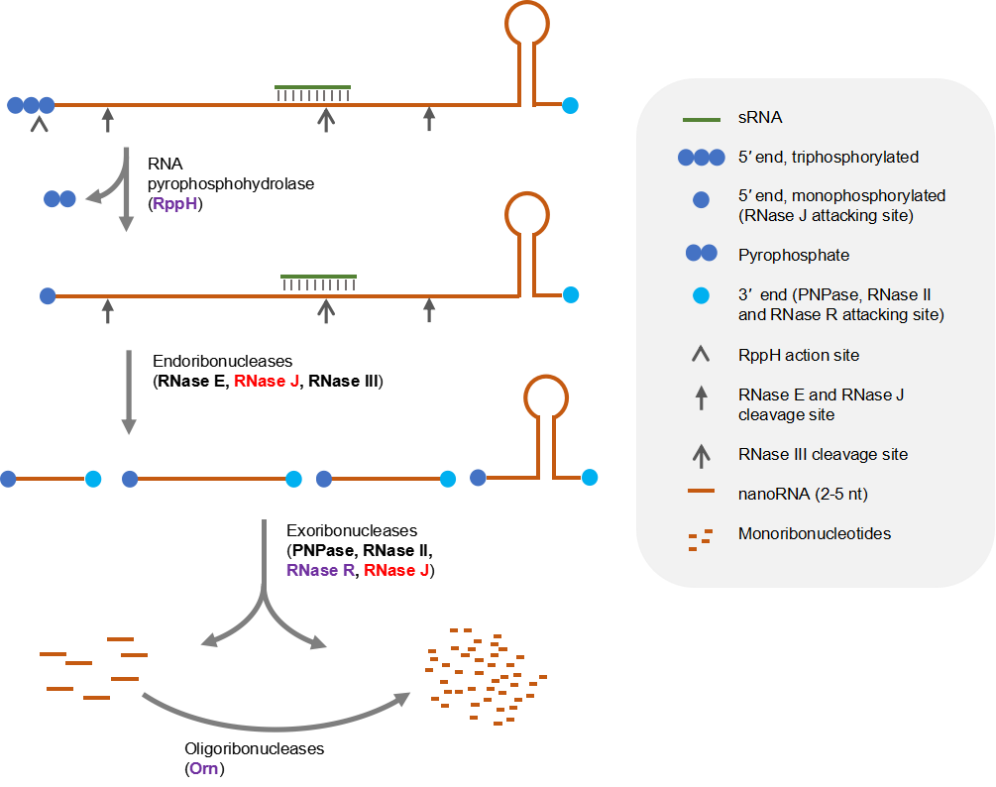
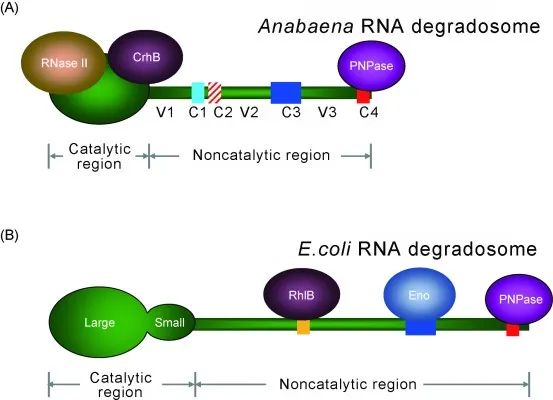
图1:细菌中RNA主要降解过程。全长RNA分子经核酸内切酶(endoribonucleases)切为较短的片段,这些短片段进一步被核酸外切酶(exoribonucleases)降解为单核苷酸和长2-5碱基的微核苷酸(nanoRNA)产物。微核苷酸新一步被寡核苷酸酶(oligoribonucleases)降解为单核苷酸,最终完成RNA的彻底降解。需要注意的是,许多带有5’-三磷酸的RNA需要先被RNA焦磷酸酶(RNA pyrophosphohydrolase)转变为5’-单磷酸RNA才能被RNA内切酶有效切割。括号内列出的是目前蓝细菌和E. coli中发现的参与RNA降解的主要的酶,其中黑色为在蓝细菌和E. coli中均存在的酶,紫色为仅存在于E. coli中的酶,而红色为仅存在于蓝细菌中的酶。 对模式菌株的研究发现,参与RNA降解的具体酶的种类及其形成的RNA降解体可能大相庭径。例如,E. coli中内切核酸酶RNase E是最重要的RNA酶,它与外切核酸酶PNPase、解旋酶RhlB等结合形成一个RNA降解体。而B. subtilis却不含RNase E,它有一个与RNase E序列完全不同但功能类似的内切酶——RNase Y。RNase Y也与其他组分形成一个不同的RNA降解体。 蓝细菌(也称蓝藻)是一个古老的细菌类群,它们是唯一能进行产氧光合作用的原核生物,其很多种属还能够生物固氮,在地球演化以及碳氮元素循环过程中一直发挥着重要功能。此外,由于能够利用太阳光将CO2转化为有机物,它们也是极具潜力的细胞工厂。近年来,人们对蓝细菌中RNA的代谢调控机制的研究逐渐兴起。 该文对蓝细菌中RNA加工降解调控的主要研究进展进行了回顾和总结,对蓝细菌和模式菌株中RNA代谢酶及降解体类进行了系统比较。文章揭示:1)不同细菌进化出了不同的RNA代谢相关酶类,但各细菌中RNA降解的总体过程类似;2)蓝细菌中存在的RNA酶的种类和数量与已知的模式菌株中的存在显著差异;3)蓝细菌的RNA酶与模式菌株中同源蛋白的生化性质大多类似,但它们在细胞中的重要性不尽相同;4)蓝细菌与E. coli的RNA降解体都以RNase E为核心,但组装方式和组分明显不同(图2)。
|